#########1.データの取り込み#########
#分析対象データは、これと同じ
#install.packages("kernlab") ※初回のみ左記コマンドを実行
library(kernlab)
data(spam)
head(spam) #項目名を確認
#データ型を確認
for (i in 1:ncol(spam)) {
print(c(names(spam[i]),class(spam[,i])))
}
#欠損値の有無
sum(is.na(spam))
#項目名の解説は、http://archive.ics.uci.edu/ml/datasets/Spambaseに掲載されている
#########2.Validate data, Train dataの生成#########
set.seed(2) #一定の結果を得るための乱数セット
trainNO <- sample(nrow(spam),nrow(spam)*0.8,replace=FALSE) #replace=TRUE:非復元一様分布からの抽出
train_dat <- spam[trainNO,]
vali_dat <- spam[-trainNO,]
#########3.train dataから決定木モデルを作成#########
#spam/nospamを予測するモデルを作成
library("rpart")
library("rpart.plot")
fit <- rpart(type~., data=train_dat,
control=rpart.control(minsplit=30,cp=0.000001), #cpの値は任意で定めた
method="class",parms=list(split="information"))
#結果を確認
par(ps=40) #フォント調整
rpart.plot(fit,type=4,extra=1)
#分岐の様子を確認し、適切なcpを確認する
par(ps=20)
printcp(fit)
plotcp(fit) #xerrorの減少幅が小さくなる箇所に注目※
#prune関数により、cpを修正することができる
#fit$cptableよりxerrorとcpの関係のテーブルを取得可能
fit$cptable
fit2 <- prune(fit, cp=fit$cptable[5,1]) #cptable[x,y]のx,yの部分は※の結果に応じて変える
#cp修正後の結果を確認
rpart.plot(fit2,type=4,extra=1)
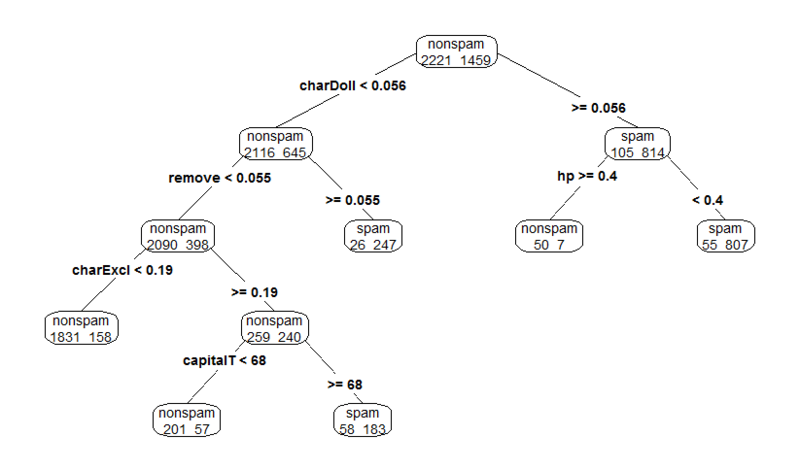
#Variable importance
attributes(fit2)
plot(fit2$variable.importance)
fit2$variable.importance
fit2
#########5.モデルをvali_datに適用し、精度検証#########
##(a)混同行列 ※分類木(目的変数がカテゴリー変数)の時のみ
predicted <- predict(fit2,newdata=vali_dat,type="class") #type="class"でtypeのspam/No spamを予測
correct <- vali_dat[,58]
(cm <- table(correct,predicted)) #全体に()を付すと表示する
(accuracyTraining <- sum(diag(cm))/sum(cm)) #精度(正解率)
(FPR <- cm[1,2]/sum(cm[1,])) #False Positive Rate
(FNR <- cm[2,1]/sum(cm[2,])) #False Negative Rate
##(b)予測値のヒストグラム
#まず、vali_datをSurvivedがYes/Noのグループに分割
vali_datNo <- subset(vali_dat, type=="nonspam")
vali_datYes <- subset(vali_dat, type=="spam")
#それぞれにモデルを適用し、spamである確率を予測
vali_datNo$P <- predict(fit2,newdata=vali_datNo,type="prob") #type="prob"でtypeのspam/No spamの確率を予測
vali_datYes$P <- predict(fit2,newdata=vali_datYes,type="prob")
#ヒストグラムを描く
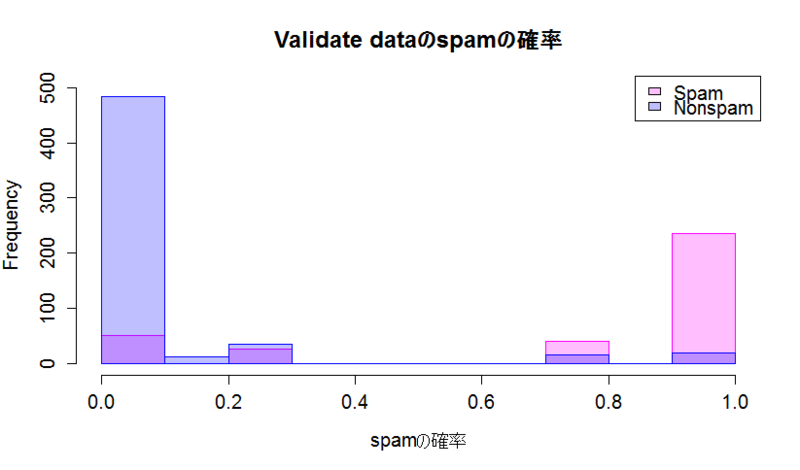
par(mfrow=c(1,1),ps=20) #グラフレイアウト調整
head(vali_datYes$P) #データの形の確認
hist(vali_datYes$P[,"spam"], col = "#ff00ff40", border = "#ff00ff", breaks = 10, xlab="spamの確率", ylim=c(0,500), main="Validate dataのspamの確率")
hist(vali_datNo$P[,"spam"], col = "#0000ff40", border = "#0000ff", breaks = 10, add = TRUE, ylim=c(0,500))
legend("topright",legend=c("Spam","Nonspam"),fill=c("#ff00ff40","#0000ff40")) # #ff00ff40,#0000ff40は半透明色
#色についてはここで紹介済み
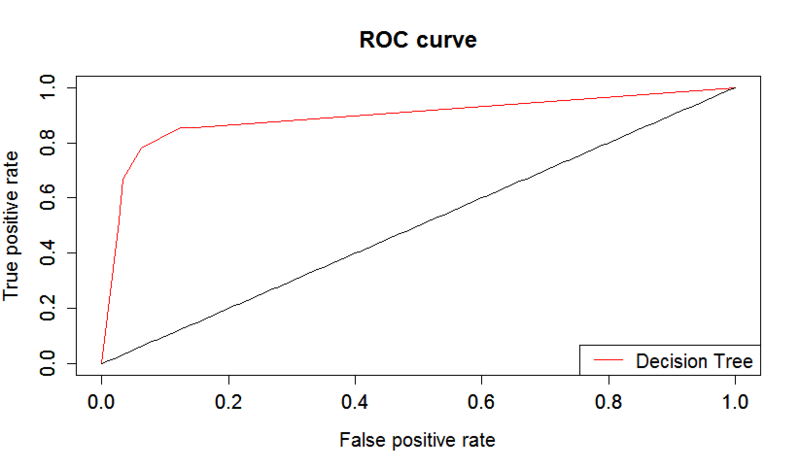
##(c)ROC/AUC
#Vali_datの予測結果を踏まえ、ROC curveを描く
par(mfrow=c(1,1)) #グラフレイアウト調整
#install.packages("ROCR") #初回のみ実行
library(ROCR)
predicted.p <- predict(fit2,newdata=vali_dat,type="prob")
predObj <- prediction(predicted.p[,2],vali_dat$type)
rocObj <- performance(predObj,measure="tpr",x.measure="fpr")
aucObj <- performance(predObj,measure="auc")
auc <- aucObj@y.values[[1]]
auc
plot(rocObj,main ="ROC curve", col="red")
par(new=T)
y <- function(x) x
plot(y,0,1,xlab="",ylab="")
legend("bottomright",legend=c("Decision Tree"),col=c("red"),lty=c(1))
##(d)R-squared
library(polycor)
predicted.p_class <- predict(fit2,newdata=vali_dat,type="class")
#カテゴリカル変数同士の相関係数を求めるには、polychorを用いる
(R <- polychor(predicted.p_class,vali_dat[,"type"],ML=TRUE)) #ML=TRUEは最尤法を意味する
(R_sq <- R^2)
#########[Additional] 6.Random Forest法及び精度の比較#########
library(randomForest)
train_rf <- randomForest(type~., data=train_dat, mtry=ncol(train_dat)-1)
print(train_rf)
attributes(train_rf)
par(ps=20) #フォント調整
plot(train_rf) #tree数を増加させていく毎にerror rateが減少する様子がわかる
head(train_rf$err.rate)
par(ps=20) #フォント調整
train_rf$importance
varImpPlot(train_rf) #Variable Improtanceのグラフ
##(a)混同行列
vali_rfp <- predict(train_rf,vali_dat[,-58])
(vali_rft<- table(vali_dat[,58], vali_rfp))
(accuracyTraining_rf <- sum(diag(vali_rft))/sum(vali_rft)) #精度(正解率)
(FPR_rf <- vali_rft[1,2]/sum(vali_rft[1,])) #False Positive Rate
(FNR_rf <- vali_rft[2,1]/sum(vali_rft[2,])) #False Negative Rate
print(c(accuracyTraining,accuracyTraining_rf)) #精度(右の数値がRandom forest)
print(c(FPR,FPR_rf)) #FPR(右の数値がRandom forest)
print(c(FNR,FNR_rf)) #FNR(右の数値がRandom forest)
##(d)R-squared
#カテゴリカル変数同士の相関係数をpolychorを用いて求める
(R_rf <- polychor(vali_dat$type,vali_rfp,ML=TRUE))
(R_rf_sq <- R_rf^2)
print(c(R_sq,R_rf_sq)) #R^2(右の数値がRandom forest)


